HMP 菌株・ゲノム標品
マイクロバイオーム標品(Microbiome Standard)
特長
- Human Microbiome Project (HMP)の標品をベースに作成
- 16S rRNA遺伝子解析やメタゲノム解析のプロトコル標準化(最適化)に
- 標品に使用している菌株や抽出ゲノムも個別にご提供
- データ解析のバイアスを除去にするため、解析ツール(One Codex社と共同開発)もご提供
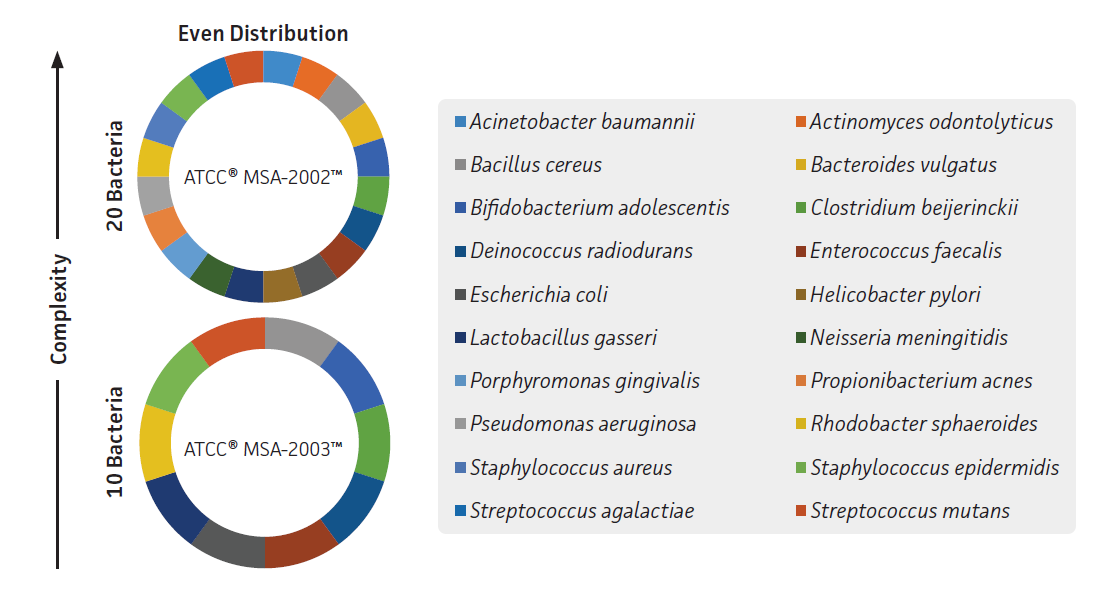
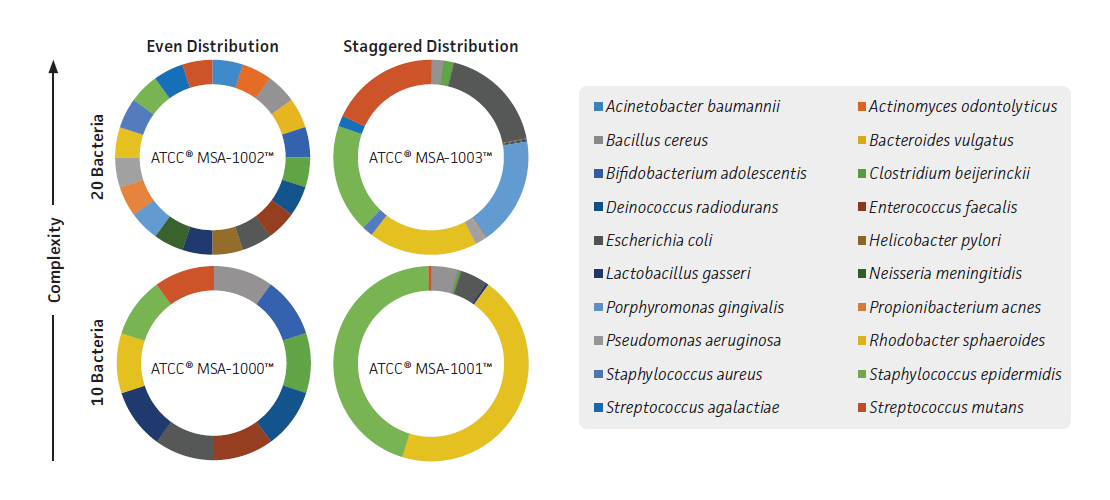
Whole Cell Microbiome Standard (菌株標品)
微生物10株及び20株を均一にミックスしたタイプをご用意しています。
各商品の菌株構成
| 20 Strain Even Mix Whole Cell Material (ATCC® MSA-2002™) | |||
|---|---|---|---|
| 構成比 | 各5% | ||
| 構成菌株 | Acinetobacter baumannii (ATCC 17978), Actinomyces odontolyticus (ATCC 17982), Bacillus cereus (ATCC 10987), Bacteroides vulgatus (ATCC 8482) Bifidobacterium adolescentis (ATCC 15703), Clostridium beijerinckii (ATCC 35702), Deinococcus radiodurans (ATCC BAA-816), Enterococcus faecalis (ATCC 47077) Escherichia coli (ATCC 700926), Helicobacter pylori (ATCC 700392), Lactobacillus gasseri (ATCC 33323), Neisseria meningitidis (ATCC BAA-335), Porphyromonas gingivalis (ATCC 33277), Propionibacterium acnes (ATCC 11828), Pseudomonas aeruginosa (ATCC 9027), Rhodobacter sphaeroides (ATCC 17029) Staphylococcus aureus (ATCC BAA-1556), Staphylococcus epidermidis (ATCC 12228), Streptococcus agalactiae (ATCC BAA-611), Streptococcus mutans (ATCC 700610) | ||
| 10 Strain Even Mix Whole Cell Material (ATCC® MSA-2003™) | |||
| 構成比 | 各10% | ||
| 構成菌株 | Bacillus cereus (ATCC 10987), Bifidobacterium adolescentis (ATCC 15703), Clostridium beijerinckii (ATCC 35702), Deinococcus radiodurans (ATCC BAA-816) Enterococcus faecalis (ATCC 47077), Escherichia coli (ATCC 700926), Lactobacillus gasseri (ATCC 33323), Rhodobacter sphaeroides (ATCC 17029) Staphylococcus epidermidis (ATCC 12228), Streptococcus mutans (ATCC 700610) | ||
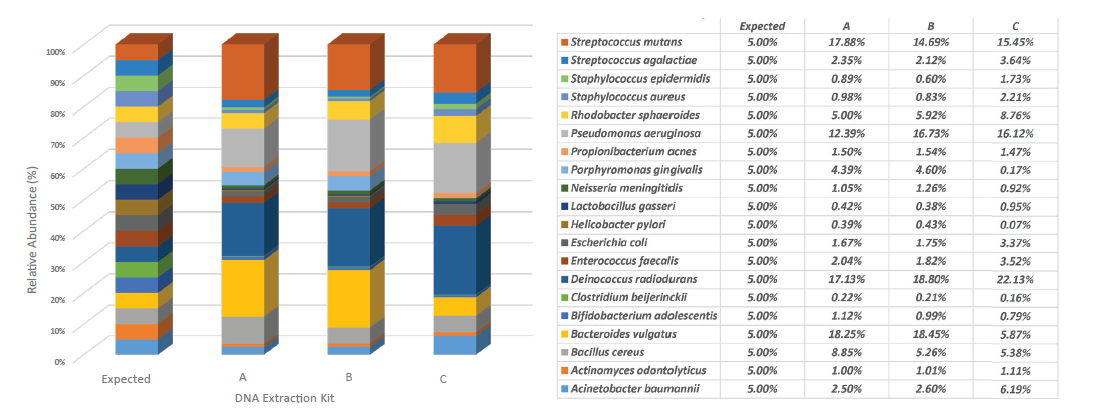
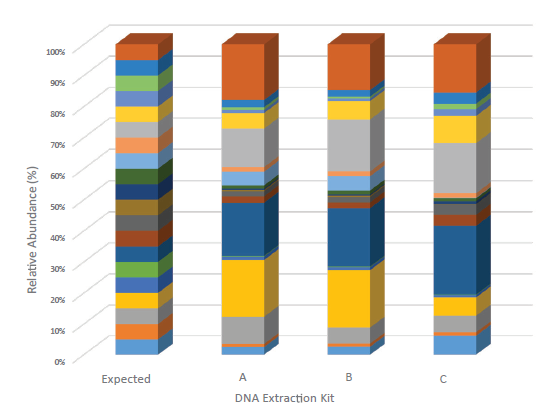
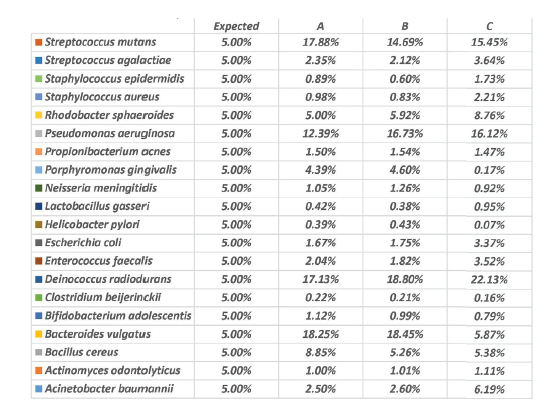
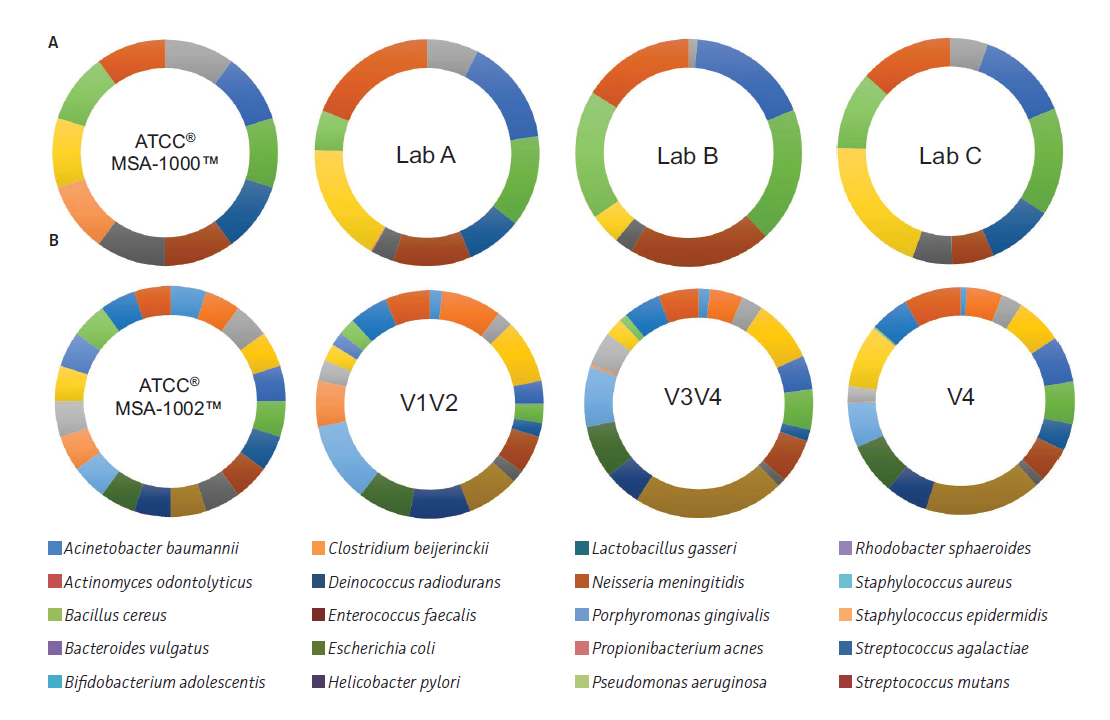
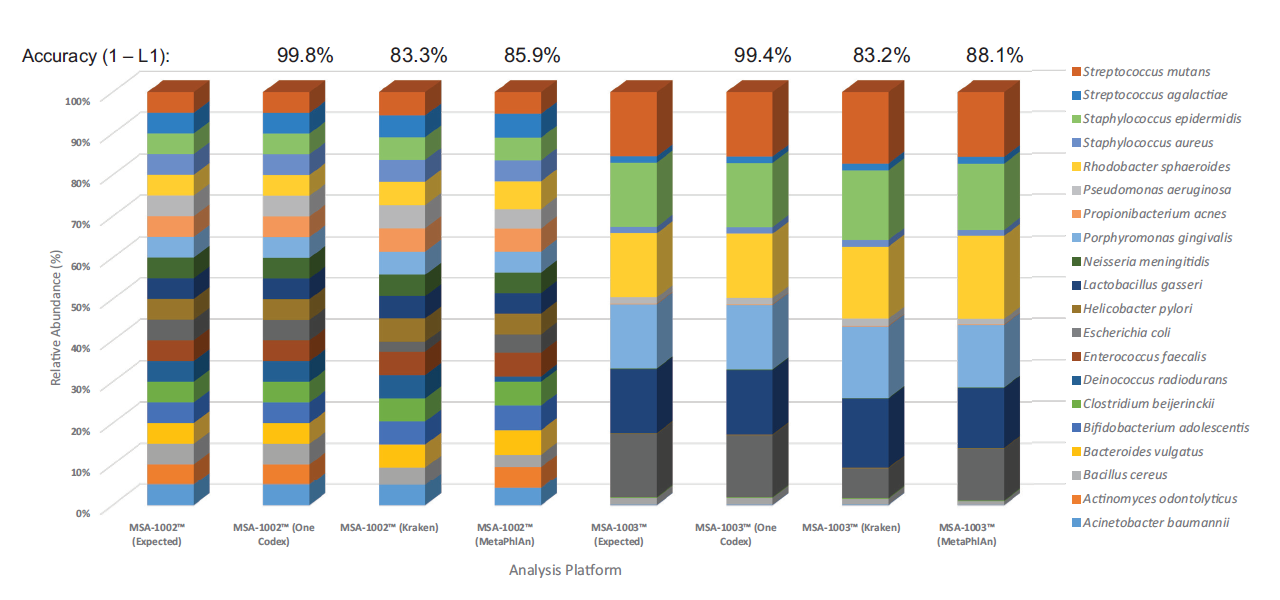
3つの異なる抽出キットを用いて解析データを比較(ATCC® MSA-2002™を使用)。Illumina社プラットフォームにて分析後、One codex社解析ツールにて解析をした。抽出キットの違いにより期待値と異なる結果となった。ゲノム抽出ステップでのプロトコル標準化(最適化)の重要性を示している。
Genomic Microbiome Standard (ゲノム標品)

微生物10株及び20株の抽出ゲノムをミックスした商品です。
均一もしくは不均一にミックスしたタイプをそれぞれご用意しています。
各商品の菌株構成
| 10 Strain Even Mix Genomic Material (ATCC® MSA-1000™) | |||
|---|---|---|---|
| 構成比 | 各10% | ||
| 構成菌株 | Bacillus cereus (ATCC 10987), Bifidobacterium adolescentis (ATCC 15703), Clostridium beijerinckii (ATCC 35702), Deinococcus radiodurans (ATCC BAA-816), Enterococcus faecalis (ATCC 47077), Escherichia coli (ATCC 700926), Lactobacillus gasseri (ATCC 33323), Cereibacter sphaeroides (ATCC 17029), Staphylococcus epidermidis (ATCC 12228), Streptococcus mutans (ATCC 700610) | ||
| 10 Strain Staggered Mix Genomic Material (ATCC® MSA-1001™) | |||
| 構成比 | 下記に記載 | ||
| 構成菌株 | 4.48% Bacillus cereus (ATCC 10987), 0.04% Bifidobacterium adolescentis (ATCC 15703), 0.45% Clostridium beijerinckii (ATCC 35702), 0.04% Deinococcus radiodurans (ATCC BAA-816), 0.04% Enterococcus faecalis (ATCC 47077), 4.48% Escherichia coli (ATCC 700926), 0.45% Lactobacillus gasseri (ATCC 33323), 44.78% Rhodobacter sphaeroides (ATCC 17029), 44.78% Staphylococcus epidermidis (ATCC 12228), 0.45% Streptococcus mutans (ATCC 700610) | ||
| 20 Strain Even Mix Genomic Material (ATCC® MSA-1002™) | |||
| 構成比 | 各5% | ||
| 構成菌株 | Acinetobacter baumannii (ATCC 17978), Actinomyces odontolyticus (ATCC 17982), Bacillus cereus (ATCC 10987), Bacteroides vulgatus (ATCC 8482), Bifidobacterium adolescentis (ATCC 15703), Clostridium beijerinckii (ATCC 35702), Deinococcus radiodurans (ATCC BAA-816), Enterococcus faecalis (ATCC 47077), Escherichia coli (ATCC 700926), Helicobacter pylori (ATCC 700392), Lactobacillus gasseri (ATCC 33323), Neisseria meningitidis (ATCC BAA-335), Porphyromonas gingivalis (ATCC 33277), Propionibacterium acnes (ATCC 11828), Pseudomonas aeruginosa (ATCC 9027), Rhodobacter sphaeroides (ATCC 17029), Staphylococcus aureus (ATCC BAA-1556), Staphylococcus epidermidis (ATCC 12228), Streptococcus agalactiae (ATCC BAA-611), Streptococcus mutans (ATCC 700610) | ||
| 20 Strain Staggered Mix Genomic Material (ATCC® MSA-1003™) | |||
| 構成比 | 下記に記載 | ||
| 構成菌株 | 0.18% Acinetobacter baumannii (ATCC 17978), 0.02% Actinomyces odontolyticus (ATCC 17982), 1.80% Bacillus cereus (ATCC 10987), 0.02% Bacteroides vulgatus (ATCC 8482), 0.02% Bifidobacterium adolescentis (ATCC 15703), 1.80% Clostridium beijerinckii (ATCC 35702), 0.02% Deinococcus radiodurans (ATCC BAA-816) 0.02% Enterococcus faecalis (ATCC 47077), 18.0% Escherichia coli (ATCC 700926), 0.18% Helicobacter pylori (ATCC 700392), 0.18% Lactobacillus gasseri (ATCC 33323) 0.18% Neisseria meningitidis (ATCC BAA-335), 18.0% Porphyromonas gingivalis (ATCC 33277), 0.18% Propionibacterium acnes (ATCC 11828), 1.80% Pseudomonas aeruginosa (ATCC 9027), 18.0% Rhodobacter sphaeroides (ATCC 17029), 1.80% Staphylococcus aureus (ATCC BAA-1556), 18.0% Staphylococcus epidermidis (ATCC 12228), 1.80% Streptococcus agalactiae (ATCC BAA-611), 18.0% Streptococcus mutans (ATCC 700610) | ||
| 20 Strain Staggered Mix Genomic Material (ATCC® MSA-4000™) | |||
| 構成比 | 下記に記載 | ||
| 構成菌株 | 0.1% Acinetobacter baumannii strain 5377 (ATCC 17978), 0.7% Enterococcus faecalis strain V583 (ATCC 700802), 1.4% Escherichia coli strain CFT073(ATCC 700928), 14.4% Klebsiella pneumoniae subsp. pneumoniae strain MGH78578 (ATCC 700721), 28.9% Neisseria meningitidis strain FAM18 (ATCC 700532), 0.3% Pseudomonas aeruginosa strain PAO1-LAC (ATCC 47085), 0.7% Staphylococcus aureus subsp. aureus (MRSA) strain FPR3757 (ATCC BAA-1556), 14.4% Staphylococcus aureus subsp. aureus (MSSA) strain TCH959 (ATCC BAA-1718), 2.9% Streptococcus agalacitae strain 2603 V/R (ATCC BAA-611), 28.9% Streptococcus pneumoniae strain Spain 23F-1 (ATCC 700669), 7.2% Streptococcus pyogenes strain SF370 (ATCC 700294) | ||
A:異なるラボ(Lab A~C)で同一プライマーを用いた際の解析データ。
(ATCC® MSA-1000™を使用)
16S rRNA V4プライマーを用いてPCR増幅を行い、それらの増幅物をblind sampleとして外部機関に解析を依頼した(lllumina社プラットフォームにより標準16Sプロトコル[Earth Microbiome Project]を使用)。同一プライマーを使用しているが、期待値と大きく異なる結果となった。
B:異なるプライマーを用いた際の解析データ。(ATCC® MSA-1002™を使用)
異なるプライマー(V1V2, V3V4及びV4領域をカバーするよう設計)によりPCR増幅を行い、Illumina社プラットフォームにより分析し、One Codex社解析ツールにて解析した。プライマーの設計箇所の違いにより期待値と異なる結果となった。
これらの結果はいずれもプロトコル標準化(最適化)の必要性を示している。
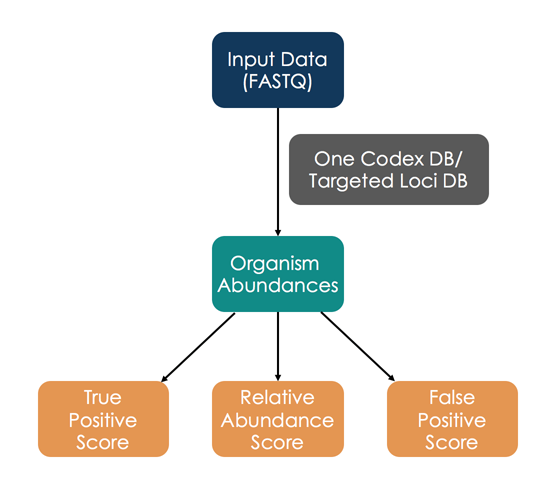
State-of-the art Bioinfomatics Tool
(One Codex社と共同開発した解析ツール)

*標品購入者のみ使用できる専用の解析ツールです。
特長
- NGSデータ(FASTQ)のアップロード及び各種設定のみの簡便な仕様
- 16S rRNA遺伝子解析データ及びショットガンシークエンスデータが使用可能
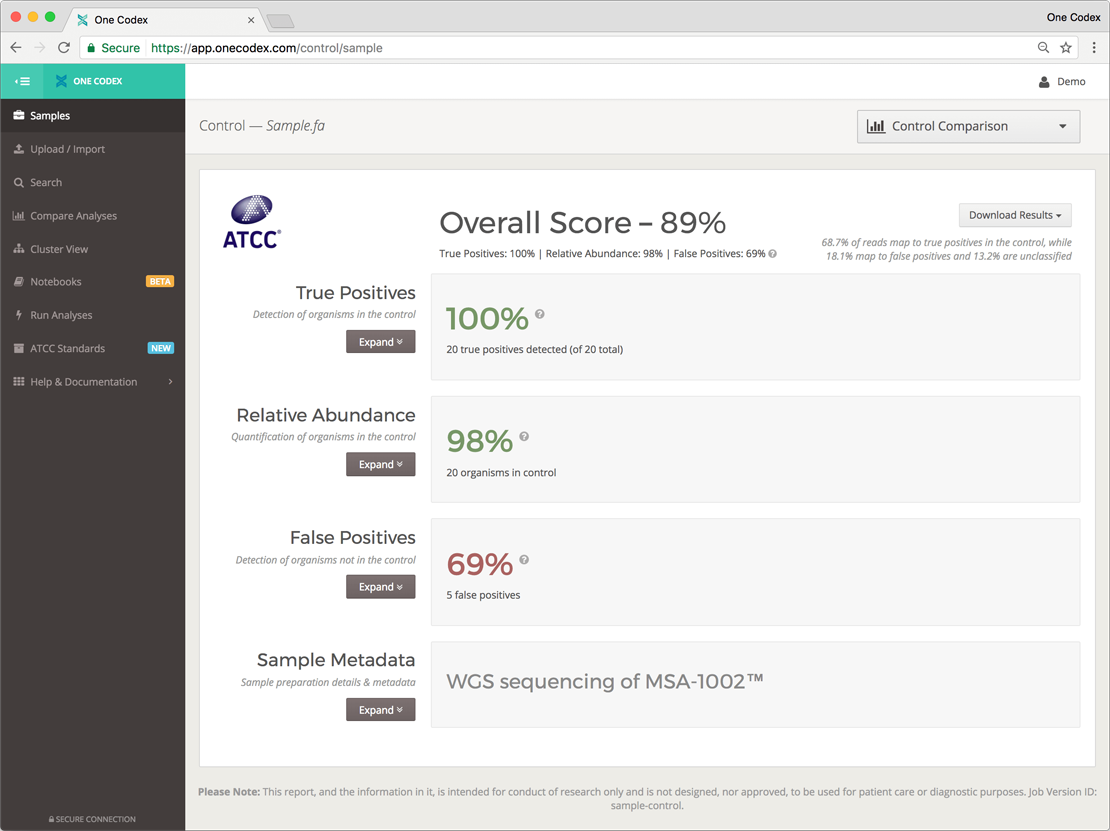
- 各微生物の相対値、true positive及びfalse positiveをスコア化
NGSデータ(FASTQ)をOne Codex社ATCC標品専用解析ページよりアップロードし、各種設定を行うのみで簡便にご使用頂けます。
解析データ例:https://app.onecodex.com/control/sample
解析ツールの使用方法はこちら
 創薬支援サービスはこちら細胞・ヒト組織商材についてはこちらのページで紹介しております。
創薬支援サービスはこちら細胞・ヒト組織商材についてはこちらのページで紹介しております。